Produktinformation
Verfügbar als maßgeschneidertes Produkt nach Ihren Bedürfnissen hergestellt: Von der Lieferung in großen Mengen und kundenspezifischen Chargengrößen bis hin zum selbst hergestellten Produkt mit der entsprechenden cfDNA im Puffer!
OEM-Optionen:
Wenn Sie Materialien als Kit-Kontrollen oder als Drittanbieter-Kontrollen für die Validierung Ihrer Kits in Kundenlabors benötigen, kontaktieren Sie uns bitte über: sales@sens-id.com
Selbst hergestellte Option:

So stellen Sie Ihr vollständiges Plasmakontrollmaterial für den Arbeitsablauf her:
Wenn Sie es vorziehen, Ihre eigenen Full-Workflow-Kontrollen herzustellen, empfehlen wir die Verwendung unserer DNA-depletierten Plasmaprodukte. Mischen Sie die DNA nach Bedarf mit dem DNA-abgereicherten Plasma, um Plasmamaterialien mit den erforderlichen DNA-Konzentrationen zu erhalten.
Dieses Produkt als cfDNA in Puffer finden Sie hier:
- EGFR-Multiplex 1% SID-000017
Kombinieren sie die cfDNA in Puffer mit:
- 5 ml human Plasma (human-tech) 1-fold concentrated
- 40 ml human Plasma (human-tech) 1-fold concentrated
- 1,000 ml human Plasma (human-tech) 1-fold concentrated
Und stellen Sie Ihre eigenen Plasmamaterialien her.
Diese Produkte sind in idealer Weise geeignet für digitale PCR und/oder Next Generation Sequencing (NGS). Insbesondere zur
– Validierung und Entwicklung von Sequenzierprotokollen (z.B. Amplicon Sequencierung) und PCR-Protokollen
– zur Bestimmung der Nachweisgrenze der Methode
– zur Extraktionskontrolle
Fragmentgröße (Peak)
~167 bp
Menge pro Einheit: nach Wunsch
Mutation:
AA Change (Cosmic ID, Mutation type, HGVS Nomenklatur, Exon)
- p.G719S (COSV51767289*, Substitution, c.2155G>A, Exon 18)
- p.E746_A750delELREA (COSV51765066*, Deletion, c.2236_2250del15, Exon 19)
- p.S752_I759delSPANKEI (COSV51774879*, Deletion, c.2254_2277del24, Exon 19)
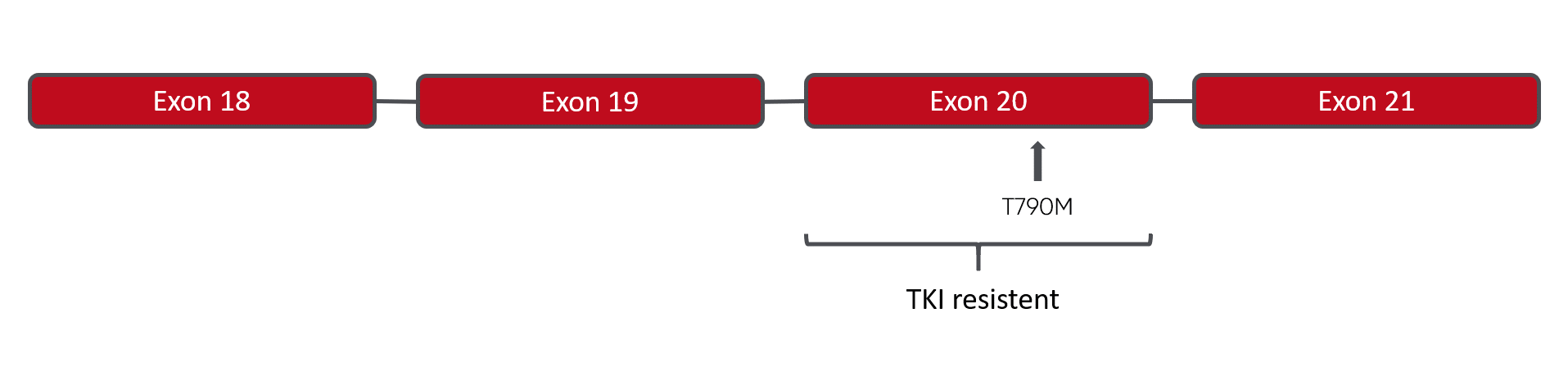
- p.S768I (COSV51768106*, Substitution, c.2303G>T, Exon 20)
- p.V769_D770insASV (COSV51850427*, Insertion, c.2303_2304ins9, Exon 20)
- p.T790M (COSV51765492*, Substitution, c.2369C>T, Exon 20)
- p.L858R (COSV51765161*, Substitution, c.2573T>G, Exon 21)
- p.L861Q (COSV51766344 *, Substitution, c.2582T>A, Exon 21)
Allelfrequenz: 1%
Puffer: humanes Plasma (human-tech)
Lagerung: 2-8 °C
Haltbarkeit:
stabil bis 24 Monate nach Herstellungsdatum (wie geliefert). Bei eigener Herstellung ist dies abhängig von Herstellbedingungen
Qualitätssicherung
Fragmentgröße: High Sensitivity DNA Kit Agilent
Allelfrequenz: dPCR
Quantifizierung:
- UV-Vis Spectrophotometry (NIST-Referenzmethode) zur Messung der gesamt DNA
- dsDNA Messung (Fluorometrisch)
Technischer Hintergrund
Ursprung: cell line GM24385 (HG002- NA24385 – huAA53E0)
Bioinformatik:
– lot specific sequencing files: LOT Search
– High-confidence variant calls: ftp://ftp-trace.ncbi.nlm.nih.gov/giab/ftp/release/AshkenazimTrio
– Raw datasets and bam files, currently including 10X Genomics, BioNano, Complete Genomics regular and LFR, 300x Illumina paired-end, Illumina 6kb mate-pair, 1000x Ion exome, custom moleculo libraries, ~0.05x Oxford Nanopore, and 70x/30x/30x PacBio: ftp://ftp-trace.ncbi.nlm.nih.gov/giab/ftp/data/AshkenazimTrio/HG002_NA24385_son/
*GRCh38 COSMICv88